Utilitzant mètodes de càlcul anteriors, el processament d’una imatge podia portar fins i tot mesos. Aquest estudi pot proporcionar una eina inestimable als biòlegs que fan recerca bàsica i també té el potencial de ser utilitzat en una gran quantitat d’aplicacions biomèdiques.
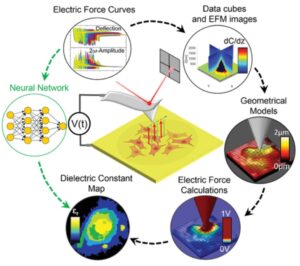
Des que Hans i Zacharias Janssen, un pare i un fill holandesos, van inventar el primer microscopi del món en 1590, la nostra curiositat pel que passa en les escales més petites ha portat al desenvolupament de dispositius cada vegada més potents. Si avancem fins a l’any 2021, no només disposem de mètodes de microscòpia òptica que ens permeten veure partícules diminutes amb més resolució que mai, sinó que també tenim tècniques no òptiques, com els microscopis de força de rastreig, amb els quals els investigadors poden construir mapes detallats d’una sèrie de propietats físiques i químiques. El grup de Caracterització Bioelèctrica a Nanoescala de l’IBEC, dirigit pel professor de la UB Gabriel Gomila, en col·laboració amb els membres del grup de Nanoscopia per la nanomedicina de l’IBEC, han estat analitzant les cèl·lules utilitzant un tipus especial de microscòpia anomenada Microscòpia Dielèctrica de Rastreig, una tècnica avançada desenvolupada en els darrers anys amb la qual poden crear mapes d’una propietat física elèctrica anomenada constant dielèctrica. Cadascuna de les biomolècules que componen les cèl·lules és a dir, lípids, proteïnes i àcids nucleics- té una constant dielèctrica diferent, de manera que un mapa d’aquesta propietat és bàsicament un mapa de la composició cel·lular.
La tècnica que han desenvolupat té un avantatge sobre l’actual mètode òptic de referència, que consisteix a aplicar un colorant fluorescent que pot alterar la cèl·lula estudiada. El seu mètode no requereix l’addició de cap agent extern potencialment pertorbador. No obstant això, l’aplicació d’aquesta tècnica requereix un complex procés de postprocessament per convertir els observables mesurats en les magnituds físiques, el que en el cas de les cèl·lules eucariotes implica enormes quantitats de temps de càlcul. De fet, es poden trigar mesos a processar una sola imatge en un ordinador d’estació de treball, ja que la constant dielèctrica s’analitza píxel a píxel utilitzant models geomètrics locals reconstruïts.
De mesos a segons
En aquest nou estudi, publicat recentment a la revista Small Methods, els investigadors van optar per una nova tècnica per accelerar el processament de les dades del microscopi. Aquesta vegada, van utilitzar algoritmes d’aprenentatge automàtic en lloc de mètodes computacionals convencionals. El resultat va ser dràstic: un cop entrenat, l’algoritme d’aprenentatge automàtic va ser capaç de produir un mapa de composició bioquímica dielèctrica de les cèl·lules en tan sols uns segons. En l’estudi no es van afegir substàncies externes a la mostra, un objectiu llargament buscat en l’obtenció d’imatges de composició en biologia cel·lular. Van aconseguir aquests ràpids resultats utilitzant un potent tipus d’algorisme anomenat xarxes neuronals, que imita la manera en què operen les neurones de cervell humà.
L’estudi va ser descrit per Martí Checa, que el va portar a terme com a part del seu doctorat en el grup de Gomila a l’IBEC. Ara és investigador postdoctoral a l’Institut Català de Nanociència i Nanotecnologia (ICN2). “És un dels primers estudis que proporciona un mapa de composició bioquímica de cèl·lules eucariotes seques tan ràpid i sense etiquetes”, explica Checa. De fet, en aquest estudi de prova de concepte, els investigadors van utilitzar cèl·lules seques, per evitar els enormes efectes de l’aigua en els mesuraments dielèctrics per la seva elevada constant dielèctrica. En un estudi de seguiment publicat recentment, també van analitzar cèl·lules fixes en el seu estat natural en líquid. Aquí van poder comparar els valors obtinguts en els models sec i líquid per obtenir un mapa precís de les biomolècules que componen les cèl·lules eucariotes. Es tracta de les cèl·lules multiestructurades de les quals es componen els animals, plantes, fongs i altres organismes. “El següent pas en aquesta investigació és aplicar el mètode a cèl·lules vives elèctricament excitables, com les neurones, on es produeix una intensa activitat elèctrica. Ens entusiasma veure el que es pot obtenir amb la nostra tècnica en aquests sistemes”, afegeix el professor Gomila.

Imatge: Mapa de composició bioquímica dielèctrica de cèl·lules HeLa seques obtingudes amb el nou mètode desenvolupat de microscòpia.
Aplicacions biomèdiques
Els investigadors van validar la seva metodologia comparant els seus resultats amb fets ben coneguts sobre la composició de les cèl·lules, com la naturalesa rica en lípids de la membrana cel·lular o la gran quantitat d’àcids nucleics presents en el nucli. Amb aquest treball, han obert la possibilitat d’analitzar grans quantitats de cèl·lules en un temps rècord.
S’espera que aquest estudi proporcioni una eina indispensable per als biòlegs per dur a terme investigacions bàsiques, així com per obrir possibles aplicacions mèdiques. Per exemple, actualment s’estudien els canvis en les propietats dielèctriques de les cèl·lules com a possibles biomarcadors d’algunes malalties, com el càncer o les neurodegeneratives.
És el primer estudi que proporciona un mapa de composició bioquímica a nanoescala tan ràpid a partir de mesuraments dielèctrics de cèl·lules eucariotes seques, que clàssicament es consideren extremadament difícils de cartografiar causa de la seva complexa topografia tridimensional.
Martí Checa

Imatge: imatge de grup del professor Gabriel Gomila a l’Institut de Bioenginyeria de Catalunya
Article de referència:
Martí Checa, Ruben Millan-Solsona, Adrianna Glinkowska Mares, Silvia Pujals, and Gabriel Gomila. Fast Label-Free Nanoscale Composition Mapping of Eukaryotic Cells Via Scanning Dielectric Force Volume Microscopy and Machine Learning. Small Methods, 2021.
Aquest article també apareix a: Hot Topic: Artificial Intelligence and Machine Learning





