Con los métodos de cálculo anteriores, el procesamiento de una imagen podía llevar incluso meses. Este estudio puede proporcionar una herramienta inestimable a los biólogos que realizan investigación básica en biología celular y también tiene el potencial de ser utilizado en algunas aplicaciones biomédicas.
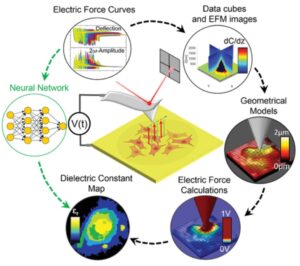
Desde que Hans y Zacharias Janssen, un padre y un hijo holandeses, inventaron el primer microscopio del mundo en 1590, nuestra curiosidad por lo que ocurre en las escalas más pequeñas ha llevado al desarrollo de instrumentos cada vez más potentes. Si avanzamos hasta el año 2021, no sólo disponemos de métodos de microscopía óptica que nos permiten ver partículas diminutas con mayor resolución que nunca, sino que también tenemos técnicas no ópticas, como los microscopios de barrido de fuerzas, con los que los investigadores pueden construir mapas detallados de una serie de propiedades físicas y químicas. El grupo de Caracterización Bioeléctrica a Nanoescala del IBEC, dirigido por el profesor de la UB Gabriel Gomila, en colaboración con los miembros del grupo de Nanoscopía para nanomedicina del IBEC, han estado analizando células utilizando un tipo especial de microscopía llamada Microscopía Dieléctrica de Barrido, una técnica avanzada desarrollada en los últimos años con la que pueden crear mapas de una propiedad física eléctrica llamada constante dieléctrica. Cada una de las biomoléculas que componen las células -es decir, lípidos, proteínas y ácidos nucleicos- tiene una constante dieléctrica diferente, por lo que un mapa de esta propiedad es básicamente un mapa de la composición celular. La ventaja principal de la técnica que han desarrollado respecto a la microscopía óptica, usada habitualmente, es que no requiere del uso de marcadores fluorescentes, los cuales pueden alterar las propiedades de la célula estudiada. Su método no requiere de la adición de ningún agente externo potencialmente perturbador. Sin embargo, la aplicación de esta técnica requiere un complejo proceso de post-procesamiento de los datos para convertir los observables medidos en las magnitudes físicas deseadas, lo que en el caso de las células eucariotas implica enormes cantidades de tiempo de cálculo. De hecho, se pueden tardar meses en procesar una sola imagen en una estación de trabajo de cálculo, ya que la constante dieléctrica se analiza píxel a píxel utilizando modelos geométricos locales reconstruidos.
De meses a segundos
En este nuevo estudio, publicado recientemente en la revista Small Methods, los investigadores optaron por una nueva técnica para acelerar el procesamiento de los datos del microscopio. Esta vez, utilizaron algoritmos de aprendizaje automático en lugar de métodos computacionales convencionales. El resultado fue drástico: una vez entrenado, el algoritmo de aprendizaje automático fue capaz de producir un mapa de composición bioquímica dieléctrica de las células en tan sólo unos segundos. En el estudio no se añadieron sustancias externas a la muestra, un objetivo largamente buscado en la obtención de imágenes de composición en biología celular. Para ello, utilizaron un potente tipo de algoritmo denominado “red neuronal”, que imita el modo en que operan las neuronas del cerebro humano.
«Es uno de los primeros estudios que proporciona un mapa de composición bioquímica de células eucariotas secas tan rápido y sin marcadores externos», explica Checa, uno de los investigadores que desarrollo el estudio y que lo llevó a cabo como parte de su doctorado en el grupo de Gomila en el IBEC, y que ahora es investigador postdoctoral en el Instituto Catalán de Nanociencia y Nanotecnología (ICN2). En este estudio de prueba de concepto, los investigadores utilizaron células secas, para evitar los enormes efectos del agua en las mediciones dieléctricas debido a su elevada constante dieléctrica. En un estudio de seguimiento publicado recientemente, los mismos autores analizaron células fijadas pero en su estado natural en medio líquido. Aquí pudieron comparar los valores obtenidos en los modelos seco y líquido para obtener un mapa preciso de las biomoléculas que componen las células eucariotas. Se trata de las células multiestructuradas de las que se componen los animales, plantas, hongos y otros organismos. «El siguiente paso en esta investigación es aplicar el método a células vivas eléctricamente excitables, como las neuronas, donde se produce una intensa actividad eléctrica. Estamos expectantes para ver lo que se puede obtener con nuestra técnica en estos sistemas», añade el profesor Gomila.

Imagen: Mapa de composición bioquímica obtenido a partir de imágenes dieléctrica de células HeLa secas obtenido con el nuevo método de microscopía desarrollado.
Aplicaciones biomédicas
Los investigadores validaron su metodología comparando sus resultados con hechos bien conocidos sobre la composición de las células, como la naturaleza rica en lípidos de la membrana celular o la gran cantidad de ácidos nucleicos presentes en el núcleo. Con este trabajo, han abierto la posibilidad de analizar grandes cantidades de células en un tiempo récord.
Se espera que este estudio proporcione una herramienta valiosa a los biólogos para llevar a cabo investigaciones básicas, así como para abrir posibles aplicaciones médicas. Por ejemplo, actualmente se estudian los cambios en las propiedades dieléctricas de las células como posibles biomarcadores de algunas enfermedades, como el cáncer o las neurodegenerativas.
Es el primer estudio que proporciona un mapa de composición bioquímica a la nanoescala tan rápido a partir de mediciones dieléctricas de células eucariotas secas, que clásicamente se consideran extremadamente difíciles de cartografiar debido a su compleja topografía tridimensional.
Martí Checa

Imagen: Foto de grupo del profesor Gabriel Gomila en el Instituto de Bioingeniería de Cataluña (IBEC).
Artículo de referencia:
Martí Checa, Ruben Millan-Solsona, Adrianna Glinkowska Mares, Silvia Pujals, and Gabriel Gomila. Fast Label-Free Nanoscale Composition Mapping of Eukaryotic Cells Via Scanning Dielectric Force Volume Microscopy and Machine Learning. Small Methods, 2021.
Este artículo también aparece en: Hot Topic: Artificial Intelligence and Machine Learning





