Un estudio dirigido por el Instituto de Bioingeniería de Cataluña (IBEC) y el Instituto de Biología Molecular de Barcelona (IBMB) ofrece la imagen más detallada hasta la fecha de NrdR, el regulador principal de las ribonucleótido reductasas (RNR) en las bacterias. Los investigadores obtuvieron las primeras imágenes detalladas de la estructura completa de la proteína NrdR y demostraron cómo los cambios en la forma y la asociación de esta proteína afectan a la forma en que controla procesos clave dentro de la célula. Los hallazgos, publicados recientemente en la revista International Journal of Biological Macromolecules, amplían nuestra comprensión de cómo las bacterias regulan la producción de los componentes moleculares del ADN, un aspecto crucial tanto para la microbiología fundamental como para el desarrollo de nuevas estrategias antimicrobianas.

Las ribonucleótido reductasas (RNR) son enzimas indispensables que convierten los ribonucleótidos en desoxirribonucleótidos (dNTP), los precursores que componen el ADN. Dado que la síntesis del ADN es fundamental para la supervivencia celular, la actividad de las RNR debe controlarse de manera muy estricta. En las bacterias, este control lo ejerce un regulador transcripcional especializado, el NrdR, que no tiene equivalente en los organismos eucariotas y, por lo tanto, representa una diana potencial para el desarrollo de antimicrobianos. A pesar de su papel central, la base estructural de la función de NrdR y los mecanismos por los que detecta los niveles de nucleótidos celulares y modula la expresión de las RNR solo se conocían parcialmente hasta el momento.
Ahora, este estudio aborda esta laguna combinando la biología estructural, la caracterización biofísica y los ensayos funcionales para dibujar cómo la estructura cuaternaria de NrdR responde a diferentes estados de nucleótidos y cómo estos cambios afectan a su actividad reguladora. Los investigadores centraron su estudio en dos importantes patógenos bacterianos: Escherichia coli, un patógeno modelo clave para el estudio de la fisiología bacteriana fundamental, y Pseudomonas aeruginosa, un patógeno oportunista reconocido por su resistencia inherente a muchos antibióticos y su papel en las infecciones crónicas. El trabajo de investigación fue dirigido por el grupo Infecciones Bacterianas: Terapias Antimicrobianas, del Instituto de Bioingeniería de Cataluña (IBEC) y el grupo de Biología Estructural de Macromoléculas Mitocondriales del Instituto de Biología Molecular de Barcelona (IBMB-CSIC), con la participación del grupo de Caracterización Bioeléctrica a Nanoescala del IBEC.
NrdR como nueva posible diana contra las infecciones bacterianas
Apuntar a un regulador tan importante podría debilitar las bacterias patógenas o ayudar a restaurar su susceptibilidad a los antibióticos existentes, lo que representa una vía prometedora para contrarrestar la creciente resistencia a los antimicrobianos.
Eduard Torrents
La caracterización exhaustiva de la estructura y el mecanismo de NrdR supone un avance significativo en nuestra comprensión de la regulación transcripcional bacteriana. Al revelar cómo las bacterias regulan la síntesis de desoxirribonucleótidos en respuesta a las fluctuaciones de nucleótidos, este trabajo proporciona una base conceptual y experimental para la identificación de nuevos objetivos antibacterianos y la innovación terapéutica futura.
Entender cómo NrdR detecta los niveles de nucleótidos y controla la expresión de genes RNR esenciales proporciona un nuevo punto de partida estratégico para el desarrollo de antimicrobianos. Dado que NrdR está ausente en las células humanas y desempeña un papel fundamental en la síntesis de precursores del ADN bacteriano, los conocimientos estructurales descubiertos aquí ofrecen una base sólida para diseñar moléculas que alteren selectivamente la homeostasis de los nucleótidos bacterianos. Estos conocimientos posicionan a NrdR como una diana antimicrobiana muy interesante cuya manipulación puede inspirar estrategias de próxima generación para combatir las infecciones bacterianas resistentes.
«Apuntar a un regulador tan importante podría debilitar las bacterias patógenas o ayudar a restaurar su susceptibilidad a los antibióticos existentes, lo que representa una vía prometedora para contrarrestar la creciente resistencia a los antimicrobianos», explica Eduard Torrents, investigador principal del grupo Infecciones Bacterianas: terapias antimicrobianas del IBEC, profesor asociado de la Universidad de Barcelona (UB), miembro de ICREA Academia y autor del estudio.
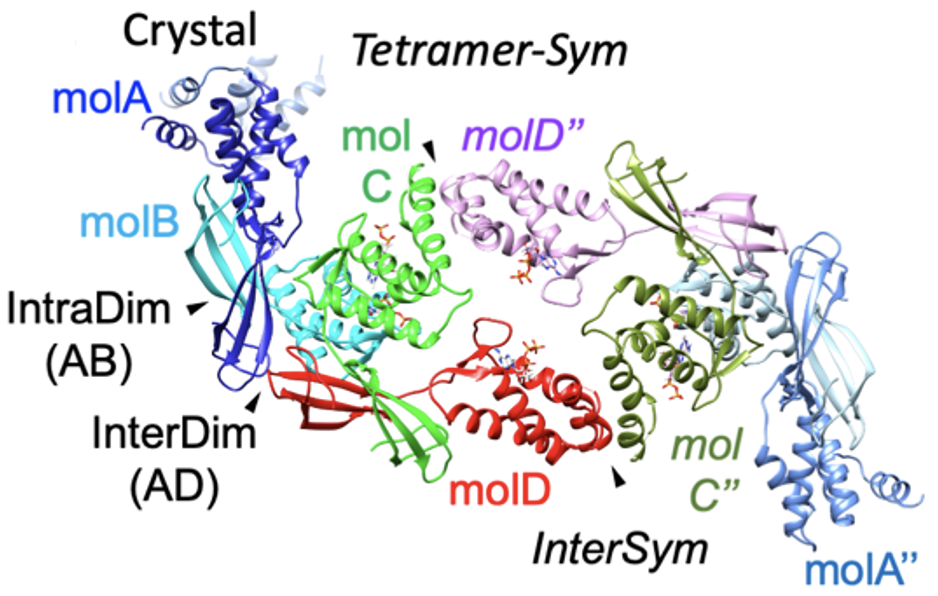
Descifrando la estructura 3D de NrdR y su importancia biológica
Los investigadores identificaron primero qué genes están controlados por NrdR en estas bacterias integrando datos transcriptómicos con análisis de motivos, lo que proporcionó una imagen más clara de cómo este regulador influye en los procesos celulares esenciales. A continuación, el equipo determinó, mediante cristalografía de proteínas con rayos X, la estructura tridimensional de la proteína NrdR en E. coli, descubriendo cómo las moléculas de proteína se asocian entre sí para formar ensamblajes. Aunque utilizaron diferentes técnicas, como la dispersión de luz multiángulo (SEC-MALS) y la microscopía de fuerza atómica, los investigadores también examinaron cómo estos ensamblajes cambian en respuesta a la presencia de diferentes nucleótidos, revelando un sistema altamente dinámico regulado por estos metabolitos. «Tras determinar la estructura cristalina, el siguiente paso crucial es descifrar si las interacciones observadas en el cristal tienen significado biológico, en este caso, cómo NrdR responde a los nucleótidos», afirma Maria Solà, investigadora principal del grupo de Biología Estructural de Macromoléculas Mitocondriales del Instituto de Biología Molecular de Barcelona (IBMB-CSIC) y autora del estudio.
Tras determinar la estructura cristalina, el siguiente paso crucial es descifrar si las interacciones observadas en el cristal tienen significado biológico, en este caso, cómo NrdR responde a los nucleótidos.
Maria Solà
Para abordar el impacto de los estados estructurales en la actividad de unión al ADN y la regulación, los investigadores realizaron una validación funcional de las interacciones proteína-proteína observadas en el cristal utilizando mutaciones puntuales, ensayos de desplazamiento de movilidad electroforética y ensayos de transcripción in vitro. Es importante destacar que el trabajo aclara cómo NrdR responde a las señales intracelulares —en particular, ATP y dATP— para cambiar entre estados activos y represivos, ajustando así la expresión de RNR de acuerdo con las necesidades celulares, explica Lucas Pedraz, co-primer autor del estudio. Estos hallazgos se oponen a las visiones anteriores, más simplistas, de la regulación de NrdR, y revelan un mecanismo finamente ajustado por el cual la unión de nucleótidos impulsa transiciones estructurales que influyen directamente en la unión al ADN y la represión génica.
En conjunto, estos resultados posicionan a NrdR como una nueva diana prometedora en la lucha contra la resistencia a los antimicrobianos, ya que ofrece una oportunidad única para interrumpir una vía reguladora esencial para la supervivencia de las bacterias, ausente en los seres humanos. Al definir la estructura de NrdR y revelar cómo gobierna la homeostasis de los nucleótidos, este trabajo no solo profundiza nuestra comprensión de la biología bacteriana, sino que también sienta una base sólida para el desarrollo de terapias de próxima generación. A medida que la resistencia a los antibióticos sigue aumentando en todo el mundo, estos conocimientos proporcionan un trampolín crucial para estrategias innovadoras que debiliten las bacterias patógenas y restauren la eficacia de los tratamientos existentes.
Artículo de referencia
Lucas Pedraz, Arkadiusz Szura, Claus Schmitz, Alba Rubio-Canalejas, Ángela Martínez-Mateos, Anthony Santella, Gabriel Gomila, Annalisa Calo, Maria Solà, Eduard Torrents. Structure and mechanistic basis of NrdR, a bacterial master regulator of ribonucleotide reduction. International Journal of Biological Macromolecules (2026). DOI: https://doi.org/10.1016/j.ijbiomac.2026.150647





